Azione 5 – Valutazione della consanguineità e della diversità genetica nelle popolazioni e calcolo dell’inbreeding
L’obiettivo è calcolare l’inbreeding e la diversità genetica in un dataset di oltre 1000 individui di 26 razze di 4 specie avicole risultante dall’unione dei genotipi ottenuti nel presente e nel precedente progetto. Piani di riduzione dell’inbreeding saranno identificati in popolazioni con eccesso di omozigosi (UniPD).
La consanguineità media, la variabilità genetica e lo stato di rischio di scomparsa saranno calcolati nelle popolazioni caratterizzate con marcatori microsatelliti. I dati saranno integrati con quelli prodotti nel progetto precedente per calcolare il contributo alla biodiversità e stimare le distanze genetiche tra razze.
Infine, si prevede di studiare la diversità genetica tra le razze rispetto ai polimorfismi del gene PAX7 (consulenza UniTO).
Per i risultati del primo TuBAvI clicca qui.
Azione 5 – TuBAvI (2017-2020): i risultati
Valutazione della consanguineità media, della variabilità genetica e del contributo di ciascuna razza alla biodiversità (UniTO)
I genotipi ottenuti in Azione 2 sono stati analizzati per la valutazione della consanguineità media, della variabilità genetica e del contributo di ciascuna razza alla biodiversità. E’ stato studiato lo stato di rischio di scomparsa e la strutturazione delle popolazioni nucleo (linee di sangue)
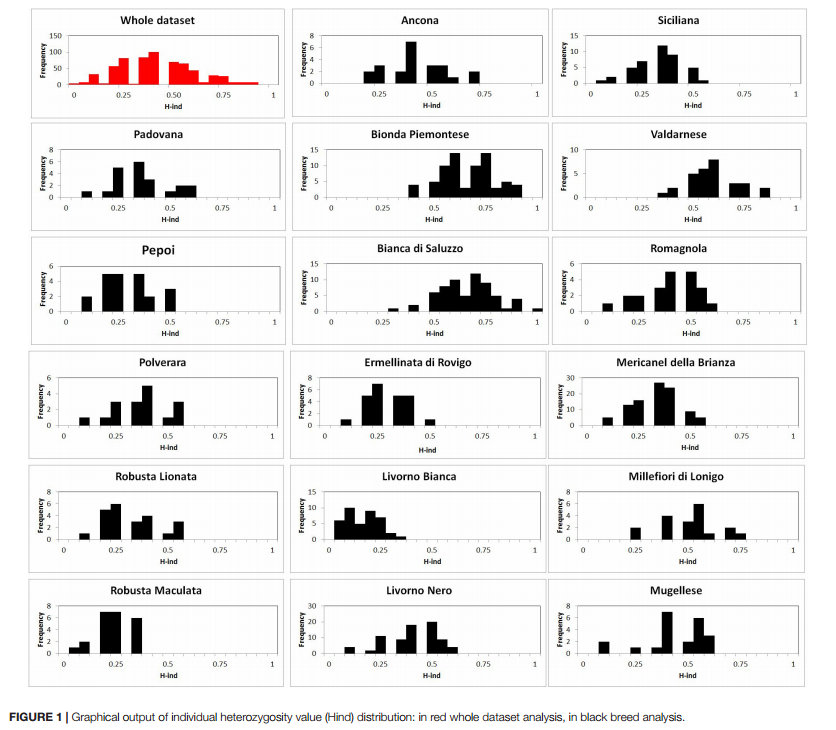
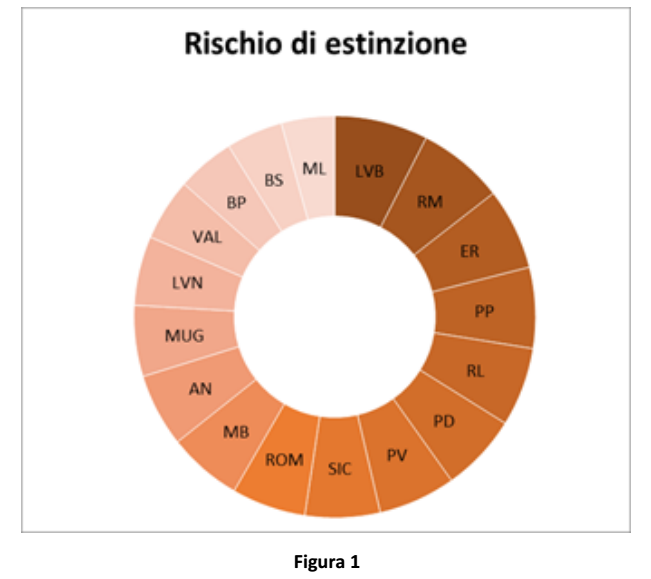
La Figura 1 mostra la distribuzione della consanguineità molecolare individuale (Eterozigosi media individuale, H-ind) nell’intera popolazione analizzata (in rosso) e delle singole popolazioni (in nero). La distribuzione complessiva segue un andamento a campana con una consanguineità del 60% nella maggior parte degli individui.
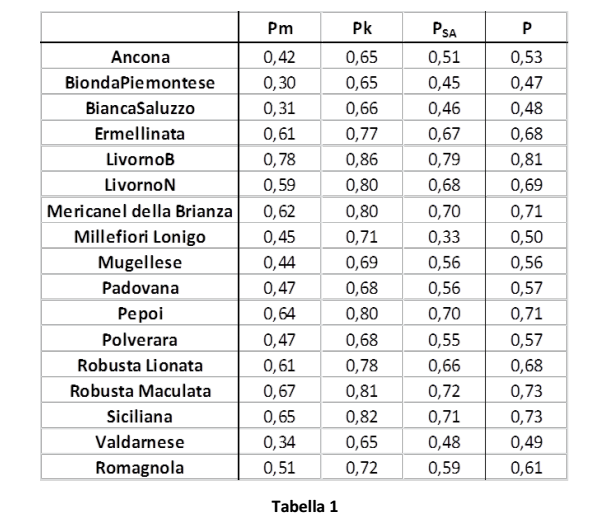
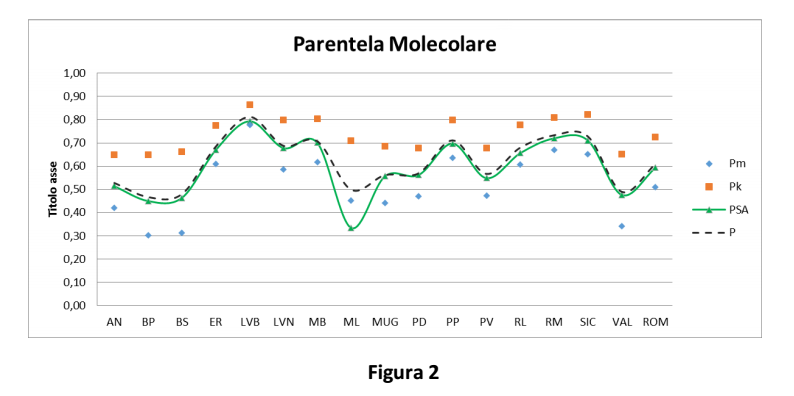
La parentela molecolare nelle popolazioni (Tabella 1 e Figura 2) è stata calcolata come media della parentela di ciascun individuo rispetto a tutti gli altri individui della stessa popolazione:
Pm: Parentela molecolare calcolata con Molkin (secondo Caballero and Toro, 2002 e Eding and Meuwissen, 2001)
Pk: Parentela calcolata sulla base delle Kinship Distance (Caballero and Toro, 2002)
PSA: Parentela calcolata sulla base del numero di alleli condivisi
P: media dei valori di parentela ottenuti con i tre approcci
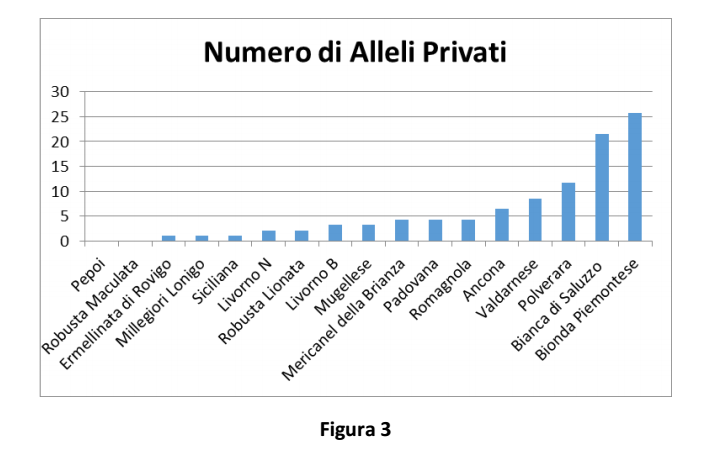
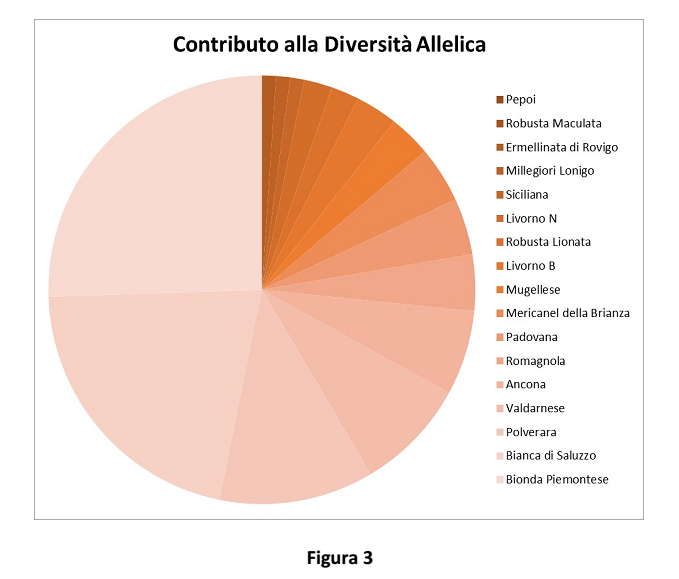
Il numero di alleli privati rappresentato in Figura 3 costituisce un indice di peculiarità della razza.
In Figura 4 sono rappresentate graficamente le distanze genetiche degli individui (DSH) appartenenti alle diverse razze. I cluster di ciascuna razza sono ben identificabili.
L’indice di Rischio di Estinzione (IRC) combina i diversi indici genetici stimati. In Figura 1, i colori più scuri indicano un rischio di estinzione più elevato.
Il contributo di ciascuna razza alla variabilità genetica della specie quale contributo alla Biodiversità è stato stimato sia come contributo alla diversità genetica globale (Figura 2), usando il metodo proposto da Caballero e Toro nel 2002, sia come contributo alla ricchezza allelica (Figura 3).