Azione 2 – Caratterizzazione genetica delle razze e delle specie autoctone
Lo studio di caratterizzazione genetica con marcatori SNP delle razze italiane di pollo viene esteso alle razze Collo nudo italiana e Millefiori piemontese. Verranno inoltre caratterizzate con marcatori SNP razze italiane di faraona (razza Camosciata), anatra (razza Mignon e Germanata veneta) e oca (razza Padovana) (UniPD). Le razze considerate nel corso dei due progetti ed i Partner coinvolti sono riportati nella Tabella sottostante.
Verranno caratterizzati polimorfismi genetici strettamente correlati con la risposta a stress da calore, per evidenziare la variabilità esistente fra diverse razze di pollo (UniPG), mentre nel tacchino la variabilità genomica e le mutazioni specifiche di ciascuna razza verranno indagate attraverso l’acquisizione di dati genomici di sequenziamento (UniMI).
Infine, la caratterizzazione con marcatori molecolari microsatelliti delle razze in conservazione presso i Partner consentirà di identificare i maschi candidati riproduttori, di monitorare la variabilità di entro razza, di individuare nuove linee familiari, di caratterizzare geneticamente nuove razze di pollo e di valutare il polimorfismo del gene PAX7, candidato ad essere utilizzato come marcatore per alcuni caratteri produttivi (consulenza UniTO).
Per i risultati del primo TuBAvI, clicca qui.
Razze autoctone di pollo, tacchino, faraona, anatra e oca sottoposte a caratterizzazione genetica
| UniMI | UniFI | UniPD | UniPG | UniPI | UniTO | |
| Razze di pollo | Mericanel della Brianza | Mugellese Valdarnese bianca |
Ermellinata di Rovigo Robusta Lionata Robusta Maculata Millefiori di Lonigo Padovana Pepoi Polverara |
Ancona Livorno |
Livorno, Siciliana | Bianca di Saluzzo Bionda Piemontese |
| Razze di tacchino | Brianzolo Bronzato comune Colli Euganei Ermellinato di Rovigo Nero d’Italia Parma e Piacenza Romagnolo |
|||||
| Razze di faraona | Camosciata | |||||
| Razze di anatra | Mignon, Germanata veneta | |||||
| Razze di oca | Padovana |
Azione 2 – TuBAvI (2017-2020): i risultati
Caratterizzazione genetica con marcatori SNP: analisi genetica di razze di pollo (UniPD)
La consistenza di una razza si valuta attraverso gli indici di diversità genetica, che sfruttano la presenza all’interno del genoma di specifici marcatori molecolari, anche detti SNP (Single-Nucleotide Polymorphisms), che consistono in una diversificazione del materiale genico a carico di un unico nucleotide, tale per cui l’allele polimorfico risulta presente nella popolazione in una proporzione superiore all’1%. Le razze analizzate presentano diversi gradi di diversità genetica, evidenziata dall’insieme degli indici stimati. La comprensione dei risultati ottenuti permette di ottenere un’indicazione sullo stato di variabilità genetica all’interno della razza e sullo stato di conservazione dell’intera popolazione.
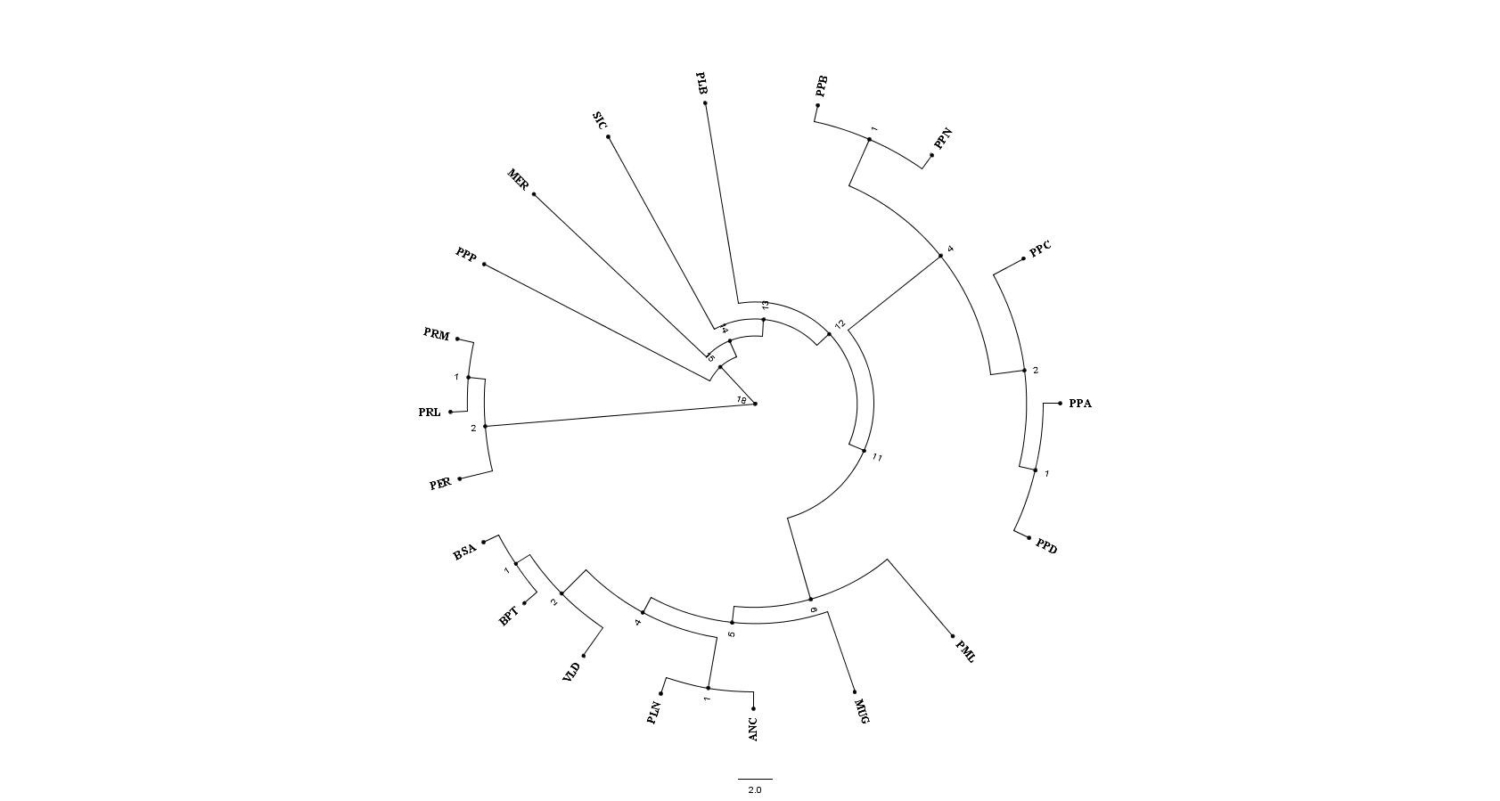
Gli studi di popolazione condotti mediante l’utilizzo di marcatori molecolari SNP permettono, inoltre, di stimare la distanza genetica delle razze con la possibilità di costruire un albero filogenetico, un diagramma che presenta le relazioni fondamentali di discendenza comune di gruppi di specie di organismi o di razze entro specie (in Figura 1 l’albero filogenetico basato sulle distanze genetiche di Raynolds fra le razze di pollo analizzate da UniPD).
Ogni nodo (o biforcazione) indica il punto in comune più recente della razza, cioè l’antenato più recente dei soggetti che si trovano ai nodi successivi. La lunghezza delle ramificazioni è invece strettamente correlata al tempo evolutivo, alla diversità genetica e ai cambiamenti genetici che intercorrono tra di essi.
Indici di diversità genetica
MAF (frequenza allelica minore): può variare da 0 a 1 ed individua il parametro di fissazione dei caratteri all’interno di una popolazione (tende a 0 se un carattere è fissato);
He (eterozigosi attesa): indica la probabilità che un individuo casualmente scelto all’interno di una popolazione, che rispetta le condizioni di equilibrio di Hardy-Weinberg, sia eterozigote per determinati caratteri; Ho (eterozigosi osservata): indica l’effettivo tasso di eterozigoti presente nella popolazione per ciascun locus allelico considerato. Ho e He permettono di analizzare la variabilità entro razza.
FHOM (coefficiente di consanguineità basato sull’eccesso di omozigosi): indica la frazione di individui di una popolazione con variante omozigote. Più tende a 1, maggiore è l’omozigosi.
ROH (Runs of homozigousity): regioni specifiche del genoma in cui si susseguono ininterrottamente marcatori SNP con genotipo omozigote. Queste porzioni di omozigosi “continua” vengono ricevute dai genitori (per discendenza mendeliana) con maggior probabilità se questi sono imparentati tra loro, pertanto hanno dimensioni e frequenza elevate nel caso in cui ci sia un’elevata consanguineità.
FROH (coefficiente di consanguineità basato sulla presenza di ROH): coefficiente di consanguineità stimato sulla presenza di regioni specifiche del genoma in continua omozigosi (ROH, Runs of homozigousity). FROH esprime la proporzione tra la lunghezza totale delle ROH di un individuo e quella del genoma coperto dagli SNP utilizzati nella caratterizzazione genetica.
Tabella 1 – Indici di diversità genetica medi per la valutazione della consistenza delle razze nell’allevamento rurale. Il numero in parentesi rappresenta il numero di animali per razza. (UniPD)
| Indici | ANC (24) |
BSA (24) |
BPT (22) |
PER (23) |
PLB (24) |
PLN (24) |
MER (23) |
PML (23) |
MUG (24) |
PPA (24) |
PPC (24) |
PPD (24) |
PPP (24) |
PPB (24) |
PPN (24) |
PRL (23) |
PRM (24) |
SIC (24) |
VLD (24) |
| MAF | 0.267 | 0.286 | 0.283 | 0.309 | 0.269 | 0.263 | 0.282 | 0.281 | 0.284 | 0.241 | 0.238 | 0.247 | 0.277 | 0.260 | 0.257 | 0.305 | 0.304 | 0.259 | 0.283 |
| He | 0.274 | 0.336 | 0.317 | 0.220 | 0.218 | 0.231 | 0.261 | 0.291 | 0.300 | 0.146 | 0.179 | 0.232 | 0.168 | 0.248 | 0.213 | 0.185 | 0.166 | 0.123 | 0.322 |
| Ho | 0.263 | 0.339 | 0.325 | 0.199 | 0.205 | 0.233 | 0.232 | 0.293 | 0.281 | 0.151 | 0.169 | 0.219 | 0.154 | 0.216 | 0.201 | 0.181 | 0.157 | 0.129 | 0.321 |
| FHOM | 0.284 | 0.076 | 0.116 | 0.459 | 0.465 | 0.365 | 0.368 | 0.166 | 0.225 | 0.588 | 0.538 | 0.404 | 0.579 | 0.411 | 0.454 | 0.508 | 0.572 | 0.648 | 0.127 |
| FROH | 0.201 | 0.081 | 0.081 | 0.305 | 0.427 | 0.296 | 0.326 | 0.202 | 0.236 | 0.509 | 0.410 | 0.230 | 0.482 | 0.310 | 0.353 | 0.353 | 0.410 | 0.607 | 0.121 |
ANC Ancona, BSA Bianca di Saluzzo, BPT Bionda Piemontese, PER Ermellinata di Rovigo, PLB Livorno Bianca, PLN Livorno Nera, MER Mericanel della Brianza, PML Millefiori di Lonigo, MUG Mugellese, PPA Padovana Argentata, PPC Padovana Camosciata, PPD Padovana Dorata, PPP Pepoi, PPB Polverara Bianca, PPN Polverara Nera, PRL Robusta Lionata, PRM Robusta Maculata, SIC Siciliana, VLD Valdarnese
Caratterizzazione genetica con marcatori SNP: analisi genetica di razze di tacchino (UniMI)
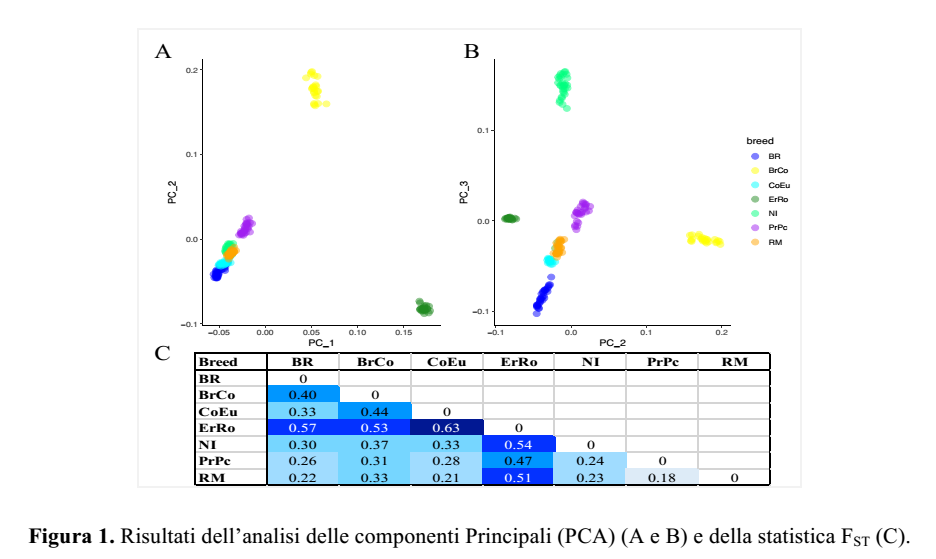
La caratterizzazione genetica di sette razze italiane di tacchino (in Tabella 1 la consistenza numerica del campione utilizzato) è stata realizzata da UniMI attraverso la genotipizzazione con chip SNP ad alta densità. I risultati della genotipizzazione hanno permesso di evidenziare la variabilità e le relazioni genetiche esistenti tra le razze analizzate. In particolare, l’analisi delle componenti principali (Figura 1A PCA_1 vs. PCA2; Figura 1B PCA_2 vs. PCA_3) mostra chiaramente come i soggetti si raggruppino in clusters corrispondenti alla razza di appartenenza. Ermellinato di Rovigo (ErRo) e Bronzato Comune (BrCo) sono le due razze che maggiormente si separano dalle altre.
Questo indica una differenza genomica marcata sia tra loro sia con le altre razze considerate, confermata anche dai valori di FST riportati in Figura 1C.
Parma e Piacenza (PrPc) e Romagnolo (RM) sono le due razze con più basso livello di differenziazione (FST = 0.18).
Per tutti i soggetti appartenenti alle razze studiate sono stati calcolati i coefficienti di consanguineità genomica FHOM e FROH. Entrambi sono risultati variabili intra e tra popolazioni e sono indicativi di un elevato livello di consanguineità (valori medi di razza riportati in Tabella 2) per le razze ErRo e CoEu e basso per la razza PrPc.
Analisi statistica: glossario
PCA (Analisi delle componenti principali): l’Analisi delle Componenti Principali (Principal Components Analysis, PCA) è una procedura di analisi statistica che consente di rappresentare la variabilità di un campione attraverso un numero ridotto di variabili altamente informative (componenti). La PCA applicata alla genomica permette di individuare cluster di razze specifici per le mutazioni individuate nel genoma delle razze studiate (le variabili).
FST: misura il grado di differenziazione delle popolazioni in base alla variazione delle frequenze alleliche tra sottopopolazioni (confronto a coppie). FST può assumere valori compresi tra 0 e 1 (FST=1 massima differenziazione; FST =0 nessuna differenziazione).